БЛАТ (биоинформатика)
BLAT ( инструмент BLAST -подобного выравнивания) — это алгоритм парного выравнивания последовательностей , разработанный Джимом Кентом из Калифорнийского университета в Санта-Крузе (UCSC) в начале 2000-х для помощи в сборке и аннотации генома человека . [1] Он был разработан в первую очередь для сокращения времени, необходимого для сопоставления миллионов считываний генома мыши и тегов экспрессированной последовательности с последовательностью генома человека. Инструменты выравнивания того времени не были способны выполнять эти операции таким образом, чтобы обеспечить регулярное обновление сборки генома человека. По сравнению с ранее существовавшими инструментами BLAT работал примерно в 500 раз быстрее при выполнениивыравнивания мРНК / ДНК и примерно в 50 раз быстрее при выравнивании белок /белок. [1]
BLAT — это один из множества алгоритмов, разработанных для анализа и сравнения биологических последовательностей, таких как ДНК, РНК и белки, с основной целью определения гомологии для выявления биологической функции геномных последовательностей. [2] Не гарантируется нахождение математически оптимального выравнивания между двумя последовательностями, как это делают классические алгоритмы динамического программирования Нидлмана-Вунша [3] и Смита-Уотермана [4] ; скорее, он сначала пытается быстро обнаружить короткие последовательности, которые с большей вероятностью будут гомологичными, а затем выравнивает и дополнительно расширяет гомологичные области. Это похоже на эвристический BLAST [5] [6]семейство алгоритмов, но каждый инструмент пытался решить проблему своевременного и эффективного выравнивания биологических последовательностей, используя различные алгоритмические методы. [2] [7]
BLAT можно использовать для выравнивания последовательностей ДНК, а также последовательностей белков и транслируемых нуклеотидов (мРНК или ДНК). Он предназначен для лучшей работы с последовательностями с большим сходством. Поиск ДНК наиболее эффективен для приматов, а поиск белков — для наземных позвоночных. [1] [8] Кроме того, запросы белков или транслированных последовательностей более эффективны для выявления отдаленных совпадений и межвидового анализа, чем запросы последовательностей ДНК. [9] Типичные варианты использования BLAT включают следующее:
BLAT предназначен для поиска совпадений между последовательностями длиной не менее 40 оснований, которые имеют ≥95% идентичности нуклеотидов или ≥80% идентичности транслируемого белка. [9] [10]
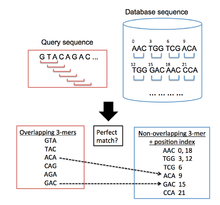
BLAT используется для поиска областей в целевой геномной базе данных, которые аналогичны исследуемой последовательности запроса. Общий алгоритмический процесс, за которым следует BLAT, аналогичен BLAST в том, что он сначала ищет короткие сегменты в базе данных и последовательностях запросов, которые имеют определенное количество совпадающих элементов. Затем эти семена выравнивания расширяются в обоих направлениях последовательностей, чтобы сформировать пары с высокими показателями. [12] Однако BLAT использует отличный от BLAST подход к индексированию, который позволяет быстро сканировать очень большие геномные и белковые базы данных на предмет сходства с последовательностью запроса. Он делает это, сохраняя индексированный список ( хэш-таблица) целевой базы данных в памяти, что значительно сокращает время, необходимое для сравнения последовательностей запросов с целевой базой данных. Этот индекс строится путем получения координат всех непересекающихся k-меров (слов с k буквами) в целевой базе данных, за исключением часто повторяющихся k-меров. Затем BLAT строит список всех перекрывающихся k-меров из последовательности запроса и ищет их в целевой базе данных, создавая список попаданий, в которых есть совпадения между последовательностями [1] (рис. 1 иллюстрирует этот процесс).
При выравнивании нуклеотидов BLAT использует третий метод, требующий двух полных совпадений слов размера 11 (11-меров). При выравнивании белков версия BLAT определяет используемую методологию поиска: когда используется версия клиент/сервер, BLAT ищет три идеальных совпадения 4-меров; когда используется автономная версия, BLAT ищет один идеальный 5-мер между последовательностями запроса и базы данных. [1]