| Сайт-специфическая ДНК-метилтрансфераза (аденин-специфическая) | ||||||||
|---|---|---|---|---|---|---|---|---|
| Идентификаторы | ||||||||
| ЕС нет. | 2.1.1.72 | |||||||
| № CAS | 69553-52-2 | |||||||
| Базы данных | ||||||||
| IntEnz | Просмотр IntEnz | |||||||
| BRENDA | BRENDA запись | |||||||
| ExPASy | Просмотр NiceZyme | |||||||
| КЕГГ | Запись в KEGG | |||||||
| MetaCyc | метаболический путь | |||||||
| ПРИАМ | профиль | |||||||
| Структуры PDB | RCSB PDB PDBe PDBsum | |||||||
| ||||||||
ДНК - аденин - метилаза , (плотина метилаза) [1] (также сайт-специфическая ДНК-метилтрансфераза (аденин-специфическая) , EC 2.1.1.72 , модификация метилаза , система рестрикция-модификация ) представляет собой фермент , который добавляет метильную группу к аденину из последовательность 5'-GATC-3 'во вновь синтезированной ДНК . [2] [3] Сразу после синтеза ДНК дочерняя цепь остается неметилированной в течение короткого времени. [4]Это орфанная метилтрансфераза, которая не является частью системы рестрикции-модификации и регулирует экспрессию генов. [5] [6] [7] [8] Этот фермент катализирует следующую химическую реакцию
- S-аденозил-L-метионин + ДНК аденин S-аденозил-L-гомоцистеин + ДНК 6-метиламинопурин
Это большая группа ферментов, уникальных для прокариот и бактериофагов. [9]
E.coli , ДНК - аденин - метилтрансфераза фермент (Р), широко используются для профилирования хроматина техники, DamID . В котором Dam сливается с представляющим интерес ДНК-связывающим белком и экспрессируется в виде трансгена в генетически поддающемся трактовке модельном организме для идентификации сайтов связывания белка. [10]
Роль в восстановлении несоответствия ДНК [ править ]
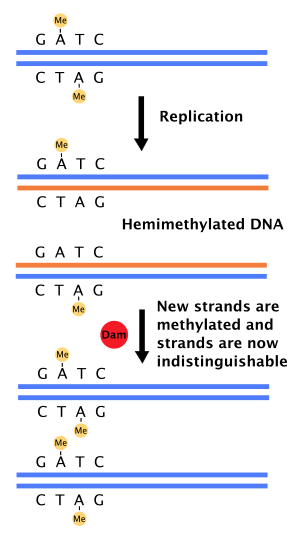
Когда ДНК - полимераза делает ошибки , что приводит к несогласованной базовой пары или небольшой вставки или удаления в процессе синтеза ДНК , то клетка будет восстановить ДНК с помощью одного из путей называется ремонта рассогласования . Однако клетка должна иметь возможность различать цепочку-матрицу и вновь синтезированную цепочку. В некоторых бактерий , нить ДНК метилируется РД метилазой, и поэтому сразу же после репликации ДНК будет полуметилированной. [4] Фермент репарации, MutS , связывается с несоответствиями в ДНК и рекрутирует MutL, который впоследствии активируетэндонуклеаза MutH. MutH связывает гемиметилированные сайты GATC и при активации избирательно расщепляет неметилированную дочернюю цепь, позволяя геликазе и экзонуклеазам вырезать зарождающуюся цепь в области, окружающей несовпадение. [4] [11] Затем цепь повторно синтезируется ДНК-полимеразой III .
Роль в регуляции репликации [ править ]
Запуск ориджина репликации (oriC) в бактериальных клетках строго контролируется, чтобы гарантировать, что репликация ДНК происходит только один раз во время каждого деления клетки. Частично это можно объяснить медленным гидролизом АТФ с помощью DnaA, белка, который связывается с повторами в oriC, чтобы инициировать репликацию. Метилаза Dam также играет роль, потому что oriC имеет 11 5'-GATC-3 'последовательностей (в E. coli ). Сразу после репликации ДНК oriC гемиметилируется и на какое-то время изолируется. Только после этого oriC высвобождается и должен быть полностью метилирован Dam-метилазой до того, как произойдет связывание DnaA.
Роль в регуляции экспрессии белков [ править ]
Dam также играет роль в продвижении и подавлении транскрипции РНК . В E. coli нижестоящие последовательности GATC метилированы , что способствует транскрипции. Например, вариация фазы пилей, ассоциированных с пиелонефритом (PAP), у уропатогенных E. coli контролируется Dam посредством метилирования двух сайтов GATC, проксимальных и дистальных по отношению к промотору PAP . [12] Учитывая его роль в регуляции белка в E. coli , ген метилазы Dam несущественен, поскольку нокаут этого гена по-прежнему оставляет бактерии жизнеспособными. [13] Сохранение жизнеспособности несмотря наНокаут гена dam также наблюдается у Salmonella и Aggregatibacter actinomycetemcomitans . [14] [15] Однако у таких организмов, как Vibrio cholerae и Yersinia pseudotuberculosis , ген dam необходим для жизнеспособности. [16] Нокаут гена dam в Aggregatibacter actinomycetemcomitans приводил к нарушению регуляции уровня белка, лейкотоксина, а также снижал способность микробов вторгаться в эпителиальные клетки полости рта. [15] Дополнительно было проведено исследование Streptococcus mutans с дефицитом метилазы дам., стоматологический патоген, выявил нарушение регуляции 103 генов, некоторые из которых обладают кариесогенным потенциалом. [16]
Структурные особенности [ править ]
Сходство каталитических доменов C5-цитозинметилтрансфераз и N6- и N4-аденин-метилтрансфераз вызвало большой интерес в понимании основ функциональных сходств и различий. Метилтрансферазы или метилазы классифицируются на три группы (группы α, β и γ) на основе последовательного порядка определенных 9 мотивов и целевого домена распознавания (TRD). [17] Мотив I состоит из трипептида Gly-X-Gly и называется G-петлей и участвует в связывании кофактора S-аденозилметионина . [18] Мотив II является высококонсервативным среди N4- и N6-аденинметилаз и содержит отрицательно заряженную аминокислоту, за которой следует гидрофобная боковая цепь в последних положениях β2-цепи для связывания AdoMet.. [17] Мотив III также участвует в связывании Adomet. Мотив IV особенно важен и хорошо известен при характеристике метилазы. Он состоит из дипролильного компонента и является высококонсервативным среди N6-аденинметилтрансфераз в качестве мотива DPPY, однако этот мотив может варьироваться для N4-аденин- и C5-цитозинметилтрансфераз. Было обнаружено, что мотив DPPY важен для связывания AdoMet. [19] Мотивы IV-VIII играют роль в каталитической активности, а мотивы 1-III и X играют роль в связывании кофактора. Для N6-аденин метилаз последовательный порядок этих мотивов , как , например: N-терминал - X - III - III - TRD - IV - V - VI - VII - VIII - С-концевой и Е . coli Dam следует этой структурной последовательности.[17] 2015 кристаллографии эксперимент показалчто E . coli Dam метилаза была способна связывать ДНК, не являющуюся GATC, с той же последовательностью мотивов, о которой говорилось; Авторы полагают, что полученная структура может служить основанием для репрессии транскрипции, не основанной на метилировании. [20]
Орфанные бактериальные метилазы и метилазы бактериофагов [ править ]
Дам-метилаза - это орфанная метилтрансфераза, которая не является частью системы рестрикции-модификации, но действует независимо, регулируя экспрессию генов, репарацию несовпадений и репликацию бактерий среди многих других функций. Это не единственный пример орфанской метилтрансферазы, поскольку существует регулируемая клеточным циклом метилтрансфераза (CcrM), которая метилирует полу-метилированную ДНК 5'-GANTC-'3 для контроля жизненного цикла Caulobacter crescentus и других родственных видов. [21]
В отличие от своих бактериальных аналогов, фаговые орфанные метилтрансферазы также существуют, особенно в T2, T4 и других T-четных бактериофагах, которые инфицируют E. coli. [5] В ходе исследования было выявлено, что, несмотря на общую гомологию последовательностей, аминокислотные последовательности метилазы E. coli и T4 Dam имеют идентичность последовательностей до 64% в четырех областях длиной от 11 до 33 остатков, что предполагает общее эволюционное происхождение. для генов бактериальной и фаговой метилазы. [22] Метилазы Т2 и Т4 отличаются от метилазы E. coli Dam не только своей способностью метилировать 5-гидроксиметилцитозин, но также метилировать неканонические участки ДНК. Несмотря на обширные исследования in vitroхарактеристика этих избранных фаговых орфанных метилтрансфераз, их биологическое назначение до сих пор неясно. [5]
См. Также [ править ]
- ДНК-метилтрансфераза
- Метилирование ДНК
- Репликация ДНК
Ссылки [ править ]
- ^ Браун, Теренс (2002). «Глава 14: Мутация, восстановление и рекомбинация. Раздел 2.3». Геномы . Наука о гирляндах. ISBN 0-471-25046-5.
- Перейти ↑ Marinus MG, Morris NR (июнь 1973). «Выделение мутантов метилазы дезоксирибонуклеиновой кислоты Escherichia coli K-12» . Журнал бактериологии . 114 (3): 1143–50. DOI : 10.1128 / JB.114.3.1143-1150.1973 . PMC 285375 . PMID 4576399 .
- ^ Гайер GE, Modrich P (февраль 1979). «Последовательность распознавания метилазы dam из Escherichia coli K12 и способ расщепления эндонуклеазой Dpn I». Журнал биологической химии . 254 (4): 1408–13. PMID 368070 .
- ^ a b c Баррас F, Маринус MG (1989). «Великий GATC: метилирование ДНК в E. coli». Тенденции в генетике . 5 (5): 139–143. DOI : 10.1016 / 0168-9525 (89) 90054-1 . PMID 2667217 .
- ^ a b c Мерфи Дж., Махони Дж., Эйнсворт С., Наута А., Ван Синдерен Д. (декабрь 2013 г.). "Бактериофаги-орфанные ДНК-метилтрансферазы: понимание их бактериального происхождения, функции и возникновения" . Прикладная и экологическая микробиология . 79 (24): 7547–55. DOI : 10,1128 / aem.02229-13 . PMC 3837797 . PMID 24123737 .
- Перейти ↑ Kessler C, Manta V (август 1990 г.). «Специфичность рестрикционных эндонуклеаз и модификации ДНК метилтрансфераз - обзор (издание 3)». Джин . 92 (1–2): 1–248. DOI : 10.1016 / 0378-1119 (90) 90486-B . PMID 2172084 .
- ^ Робертс RJ (апрель 1990). «Рестрикционные ферменты и их изошизомеры» . Исследования нуклеиновых кислот . 18 Дополнение: 2331–65. DOI : 10.1093 / nar / 18.suppl.2331 . PMC 331877 . PMID 2159140 .
- Перейти ↑ Yuan R (1981). «Структура и механизм многофункциональных эндонуклеаз рестрикции». Ежегодный обзор биохимии . 50 : 285–319. DOI : 10.1146 / annurev.bi.50.070181.001441 . PMID 6267988 .
- ^ Робертс RJ, Macelis D (ред.). «База данных рестрикционных ферментов» . REBASE . Проверено 22 фев 2020 .
- ^ Aughey GN, Саутолл TD (январь 2016). «Черт возьми, это хорошо! DamID-профилирование белок-ДНК взаимодействий» . Междисциплинарные обзоры Wiley. Биология развития . 5 (1): 25–37. DOI : 10.1002 / wdev.205 . PMC 4737221 . PMID 26383089 .
- ^ Løbner-Olesen A, Skovgaard O, Мэринус MG (апрель 2005). «Метилирование плотины: координация клеточных процессов». Текущее мнение в микробиологии . 8 (2): 154–60. DOI : 10.1016 / j.mib.2005.02.009 . PMID 15802246 .
- ^ Casadesús J, Low D (сентябрь 2006). «Эпигенетическая регуляция генов в бактериальном мире» . Обзоры микробиологии и молекулярной биологии . 70 (3): 830–56. DOI : 10.1128 / MMBR.00016-06 . PMC 1594586 . PMID 16959970 .
- ^ Bale A, d'Alarcao M, Мэринус MG (февраль 1979). «Характеристика мутантов по метилированию аденина ДНК Escherichia coli K12». Мутационные исследования . 59 (2): 157–65. DOI : 10.1016 / 0027-5107 (79) 90153-2 . PMID 375073 .
- Перейти ↑ Nicholson B, Low D (февраль 2000 г.). «Зависимая от метилирования ДНК регуляция экспрессии pef в Salmonella typhimurium» . Молекулярная микробиология . 35 (4): 728–42. DOI : 10.1046 / j.1365-2958.2000.01743.x . PMID 10692151 .
- ^ a b Wu H, Lippmann JE, Oza JP, Zeng M, Fives-Taylor P, Reich NO (август 2006 г.). «Инактивация ДНК-аденинметилтрансферазы изменяет факторы вирулентности Actinobacillus actinomycetemcomitans». Микробиология и иммунология полости рта . 21 (4): 238–44. DOI : 10.1111 / j.1399-302x.2006.00284.x . PMID 16842508 .
- ^ a b Хулио С.М., Хейтофф Д.М., Провенцано Д., Клозе К.Э., Синшеймер Р.Л., Низкий Д.А., Махан М.Дж. (декабрь 2001 г.). «ДНК-аденинметилаза необходима для жизнеспособности и играет роль в патогенезе Yersinia pseudotuberculosis и Vibrio cholerae» . Инфекция и иммунитет . 69 (12): 7610–5. DOI : 10.1128 / iai.69.12.7610-7615.2001 . PMC 98854 . PMID 11705940 .
- ^ a b c Мэлоун Т., Блюменталь Р.М., Ченг X (ноябрь 1995 г.). «Структурный анализ выявляет девять мотивов последовательности, консервативных среди ДНК-аминометилтрансфераз, и предлагает каталитический механизм для этих ферментов». Журнал молекулярной биологии . 253 (4): 618–32. DOI : 10.1006 / jmbi.1995.0577 . PMID 7473738 .
- ^ Schluckebier G, M O'Gara, Saenger W, X Cheng (март 1995). «Универсальная каталитическая доменная структура AdoMet-зависимых метилтрансфераз». Журнал молекулярной биологии . 247 (1): 16–20. DOI : 10.1006 / jmbi.1994.0117 . PMID 7897657 .
- ^ Kossykh В.Г., Schlagman SL, Hattman S (июль 1993). «Консервативный мотив последовательности DPPY в области IV ДНК- [N-аденин] -метилтрансферазы Dam фага T4 важен для связывания S-аденозил-L-метионина» . Исследования нуклеиновых кислот . 21 (15): 3563–6. DOI : 10.1093 / NAR / 21.15.3563 . PMC 331459 . PMID 16617501 .
- ^ Horton JR, Zhang X, Блюменталь RM, Cheng X (апрель 2015). «Структуры ДНК-аденин-метилтрансферазы (Dam) Escherichia coli в комплексе с последовательностью, не являющейся GATC: потенциальные последствия для независимой от метилирования репрессии транскрипции» . Исследования нуклеиновых кислот . 43 (8): 4296–308. DOI : 10.1093 / NAR / gkv251 . PMC 4417163 . PMID 25845600 .
- ^ Zweiger G, Марчиньский G, Шапиро L (январь 1994). «Метилтрансфераза ДНК Caulobacter, которая функционирует только в предделенной клетке». Журнал молекулярной биологии . 235 (2): 472–85. DOI : 10.1006 / jmbi.1994.1007 . PMID 8289276 .
- ^ Hattman S, Уилкинсон Дж, Суинтон D, Schlagman S, Макдональд ЛС, Mosig G (ноябрь 1985). «Общее эволюционное происхождение генов ДНК-аденинметилтрансферазы плотины фага Т4 и хозяина Escherichia coli dam» . Журнал бактериологии . 164 (2): 932–7. DOI : 10.1128 / JB.164.2.932-937.1985 . PMC 214344 . PMID 3902803 .
Внешние ссылки [ править ]
- http://fangman-brewer.genetics.washington.edu/hemimethylation.html