| Трансфер-мессенджер РНК | |
|---|---|
 | |
| Идентификаторы | |
| Символ | тмРНК |
| Рфам | RF00023 |
| Прочие данные | |
| Тип РНК | ген |
| Структуры PDB | PDBe |
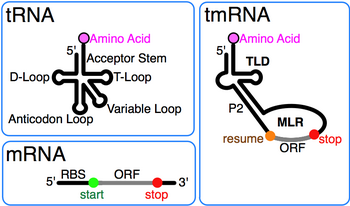
Тмрнк (сокращенно tmRNA , также известный как 10Sa РНК и ее генетическим именем SsrA ) является бактериальной РНК - молекулы с двойной тРНК -как и матричной РНК - подобных свойств. ТмРНК образует рибонуклеопротеиновый комплекс ( тмРНП ) вместе с малым белком B ( SmpB ), фактором элонгации Tu ( EF-Tu ) и рибосомным белком S1. При транс- трансляции тмРНК и связанные с ней белки связываются с бактериальными рибосомами, которые застопорились в середине биосинтеза белка., например, при достижении конца информационной РНК, которая потеряла свой стоп-кодон. ТмРНК удивительно универсальна: она рециркулирует застопорившуюся рибосому, добавляет индуцирующую протеолиз метку к незавершенному полипептиду и способствует деградации аберрантной матричной РНК . [1] У большинства бактерий эти функции выполняются стандартными цельными тмРНК . У других видов бактерий пермутированный ген ssrA продуцирует состоящую из двух частей тмРНК, в которой две отдельные цепи РНК соединены спариванием оснований.
Открытие и ранние работы [ править ]
тмРНК сначала была обозначена как 10Sa РНК после того, как смешанная электрофоретическая фракция «10S» РНК Escherichia coli была далее разделена на тмРНК и РНК РНКазы P аналогичного размера (10Sb). [2] Присутствие псевдоуридина в смешанной 10S РНК указывает на то, что тмРНК имеет модифицированные основания, обнаруженные также в тРНК . Сходство 3'-конца тмРНК с Т-стержневой петлей тРНК было впервые обнаружено при секвенировании ssrA из Mycobacterium tuberculosis . [3] Последующее сравнение последовательностей выявило полный тРНК-подобный домен (TLD), образованный 5 ' и 3'концы тмРНК, включая акцепторный стержень с элементами, подобными элементам в тРНК аланина, которые способствуют ее аминоацилированию с помощью лигазы аланин-тРНК . [4] Он также выявил отличия от тРНК : антикодоновое плечо отсутствует в тмРНК, а область D-плеча представляет собой петлю без пар оснований.
Структура [ править ]
Вторичная структура стандартных цельных тмРНК [ править ]
Полная вторичная структура тмРНК E. coli была выяснена с помощью сравнительного анализа последовательностей и структурного зондирования . [5] [6] Пары оснований Watson-Crick и GU были идентифицированы путем сравнения бактериальных последовательностей тмРНК с использованием автоматизированных вычислительных методов в сочетании с процедурами ручного выравнивания . [7] [8] На прилагаемом рисунке показан паттерн спаривания оснований этой прототипной тмРНК, которая организована в 12 филогенетически поддерживаемых спиралей (также называемых парами от P1 до P12), некоторые из которых разделены на спиральные сегменты.
Характерной особенностью каждой тмРНК является консервативный тРНК-подобный домен (TLD), состоящий из спиралей 1, 12 и 2a (аналоги акцепторной основы тРНК, Т-ствола и вариабельной основы, соответственно) и содержащий 5'-монофосфат и аланилатные 3 'концы CCA. МРНК-подобная область (MLR) в стандартной тмРНК представляет собой большую петлю, содержащую псевдоузлы и кодирующую последовательность (CDS) для пептида- метки , отмеченную кодоном возобновления и стоп-кодоном . Кодируемый пептид-метка (ANDENYALAA в E. coli ) варьируется среди бактерий, возможно, в зависимости от набора доступных протеаз и адаптеров. [9]
тмРНК обычно содержат четыре псевдоузла : один (pk1) перед теговым пептидом CDS, а другие три псевдоузла (от pk2 до pk4) ниже CDS. Области псевдоузла, хотя в целом законсервированы, эволюционно пластичны. Например, в (цельных) тмРНК цианобактерий pk4 замещен двумя тандемно расположенными более мелкими псевдоузлами. Это указывает на то, что сворачивание тмРНК вне TLD может быть важным, однако в области псевдоузла отсутствуют консервативные остатки, и псевдоузлы являются одними из первых структур, которые теряются, поскольку последовательности ssrA расходятся в клонах пластид и эндосимбионтов. Спаривание оснований в области трех псевдоузлов тмРНК E. coli нарушается во время транс- трансляции .[7] [10]
Двухкомпонентные тмРНК [ править ]
Циркулярно пермутированный ssrA был зарегистрирован в трех основных линиях: i) все альфа-протеобактерии и примитивные митохондрии протистов якобид, ii) две отдельные группы цианобактерий ( Gloeobacter и клады, содержащие Prochlorococcus и многие Synechococcus ), и iii) некоторые представители бета-протеобактерий ( Cupriavidus и некоторые Rhodocyclales). [11] [12] Все они образуют одну и ту же общую форму из двух частей (акцептор и кодирующий элемент), эквивалентную стандартной форме с надрезом после рамки считывания. Ни один из них не сохраняет более двух псевдоузлов по сравнению с четырьмя (или более) стандартными тмРНК.
Alphaproteobacteria имеют две сигнатурные последовательности: замену типичной последовательности Т-петли TΨCRANY на GGCRGUA и последовательность AACAGAA в большой петле 3´-концевого псевдоузла. В митохондриях MLR был утерян, и замечательная повторная перестановка митохондриального ssrA приводит к небольшому цельному продукту в Jakoba libera . [13]
В цианобактерии обеспечивает наиболее правдоподобный случай для эволюции переставленного гена из стандартного гена, из - за сходство замечательных последовательностей между этими двумя типами генов , как они происходят в разных Synechococcus штаммов.
обработка тмРНК [ править ]
Большинство тмРНК транскрибируются как более крупные предшественники, которые процессируются так же, как тРНК . Расщепление на 5'конца является рибонуклеазой P . [4] Множественные экзонуклеазы могут участвовать в процессинге 3´ конца тмРНК, хотя РНКаза Т и РНКаза PH являются наиболее эффективными. [14] [15] В зависимости от вида бактерий 3'-CCA либо кодируется, либо добавляется тРНК нуклеотидилтрансферазой .
Подобный процессинг на внутренних сайтах пермутированной тмРНК предшественника объясняет ее физическое расщепление на две части. Двухкомпонентные тмРНК имеют два дополнительных конца, процессинг которых необходимо учитывать. Для альфа-протеобактерий один 5´ конец является необработанным стартовым сайтом транскрипции. [16] Дальний конец 3´ в некоторых случаях может быть результатом rho-независимого завершения.
Трехмерные конструкции [ править ]
Структуры с высоким разрешением полных молекул тмРНК в настоящее время недоступны, и их может быть трудно получить из-за внутренней гибкости MLR. В 2007 году кристаллическая структура TLD Thermus thermophilus, связанного с белком SmpB, была получена с разрешением 3 Å. Эта структура показывает, что SmpB имитирует стебель D и антикодон канонической тРНК, тогда как спиральный участок 2a тмРНК соответствует вариабельному плечу тРНК. [18] крио-электронной микроскопии исследование tmRNA на ранней стадии транс - -перевод показывает пространственное соотношение между рибосомой и tmRNP (tmRNA связан с EF-Tuбелок). TLD расположен рядом с центром, ассоциированным с GTPase, в 50S субъединице рибосомы; спираль 5 и псевдоузлы pk2-pk4 образуют дугу вокруг клюва 30S субъединицы рибосомы. [19]
Транс- перевод [ править ]
Кодирование с помощью tmRNA был обнаружен в 1995 году [20] , когда Симпсон и его коллеги сверхэкспрессия цитокина мыши IL-6 в E.coli , и обнаружили множественные усеченного цитокин -derived пептиды каждый мечения карбоксильной с Термини же 11-аминокислотного остатка на расширение (А ) АНДЕНЯЛАА. За исключением N-концевого аланина , который происходит с 3'-конца самой тмРНК, эта последовательность метки была прослежена до короткой открытой рамки считывания в тмРНК E. coli . Признавая, что пептид-метка обеспечивает протеолиз , была предложена модель транс- трансляции для действия тмРНК. [21]
В то время как детали механизма транс- трансляции изучаются, все согласны с тем, что тмРНК сначала занимает пустой сайт А застопорившейся рибосомы . Затем рибосома перемещается с 3'-конца усеченной матричной РНК на кодон возобновления MLR, после чего следует стадия предрасположенности к проскальзыванию, от которой трансляция продолжается нормально до тех пор, пока не встретится стоп-кодон тмРНК в рамке считывания . Транс-трансляция важна для некоторых видов бактерий, тогда как другим бактериям требуется тмРНК для выживания в стрессовых условиях роста. [22] В зависимости от организма пептид-метка может распознаваться множеством протеаз.или протеазные адаптеры. [9]
Мобильные генетические элементы и ген тмРНК [ править ]
ssrA одновременно является мишенью для одних мобильных ДНК и "пассажиром" для других. Было обнаружено, что она прервана тремя типами подвижных элементов. С помощью различных стратегий ни один из них не нарушает функцию гена: интроны группы I удаляются путем самосплайсинга, риккетсиальные палиндромные элементы (RPE) вставляются в безвредные сайты, а геномные островки, кодирующие интегразу, расщепляют свою целевую ssrA, но восстанавливают отщепленную часть. [23] [24] [25] [26]
Номера хромосомной ssrA впервые был обнаружен в геномной обследования mycobacteriophages (в 10% фагов). [27] Были обнаружены другие мобильные элементы, включая плазмиды и геномные острова, несущие ssrA . Один интересный случай - это Rhodobacter sphaeroides ATCC 17025, чей нативный ген тмРНК нарушен геномным островком; В отличие от всех других геномных островков в генах тмРНК (или тРНК) этот остров инактивировал нативный ген-мишень без восстановления, но компенсирует это за счет несения собственного гена тмРНК. Очень необычный родственник ssrA обнаружен в литическом микобактериофаге DS6A, который кодирует немного больше, чем TLD.
Митохондриальные тмРНК ( ген ssrA ) [ править ]
Кодируемая митохондриями структурно восстановленная форма тмРНК (мт-тмРНК) была впервые постулирована для жгутиков якобид Reclinomonas americana . [11] Впоследствии присутствие митохондриального гена ( ssrA ), кодирующего тмРНК, а также сайтов транскрипции и процессинга РНК было подтверждено для всех якобид, кроме одного . [28] [13] Функциональные доказательства, то есть т-tmRNA аминоацилирование с аланин , доступны для Jakoba Libera . [13] Совсем недавно ssrA был также идентифицирован в митохондриальных геномахоомицеты . [29] Как и в α-Proteobacteria (предки митохондрий ), мт-тмРНК представляют собой циркулярно переставленные двухкомпонентные молекулы РНК, за исключением Jakoba libera, где ген вернулся к кодированию цельной конформации тмРНК. [13]
Идентификация ssrA в митохондриальных геномах [ править ]
Гены митохондриальной тмРНК первоначально были распознаны как короткие последовательности, консервативные среди якобид и обладающие потенциалом складываться в отдельную тРНК-подобную вторичную структуру. Благодаря наличию девяти полных последовательностей мтДНК якобидов [28] и значительно улучшенному инструменту поиска ковариаций (Infernal; [30] [31] [32] ) была разработана ковариационная модель на основе митохондриальных тмРНК якобидов, которая идентифицировала митохондриальную ssrA. гены также у оомицетов . В настоящее время в общей сложности обнаружено 34 мт-тмРНК оомицетов шести родов: Albugo , Bremia ,Phytophthora , Pseudoperonospora , Pythium и Saprolegnia . Модель ковариации, построенная споследовательностямикак якобидов, так и оомицетов , теперь доступна в Rfam под названием «mt-tmRNA». [29]
Структура мт-тмРНК [ править ]
Стандартная бактериальная тмРНК состоит из тРНК (Ala) -подобного домена (позволяющего добавлять некодируемый аланин к мРНК, не имеющей стоп-кодирования) и мРНК-подобного домена, кодирующего белковый тег, который предназначен для полипептида для протеолиз. МРНК-подобный домен терялся в мт-тмРНК. Сравнительный анализ последовательностей указывает на особенности, типичные для мт-тмРНК. [29] Наиболее консервативной является первичная последовательность ствола акцептора аминоацила. Эта часть молекулы имеет неизменный остаток A в положении дискриминатора и пару GU в положении 3 (за исключением S eculamonas ecuadoriensis, имеющий пару GC); это положение является сайтом узнавания аланил-тРНК-синтазы. P2 представляет собой спираль переменной длины (от 3 до 10 пар оснований) и соответствует стеблю антикодона тРНК, но без петли антикодона (что не требуется для функции тмРНК). P2 стабилизирует тРНК-подобную структуру, но четыре нуклеотида, инвариантные для оомицетов и якобидов, предполагают дополнительную, в настоящее время неидентифицированную функцию. P3 имеет пять пар оснований и соответствует Т-плечу тРНК, но с разными консенсусными нуклеотидами как в парной области, так и в петле. Последовательность Т-петли сохраняется у оомицетов и якобидов с небольшими отклонениями (например, Saprolegnia ferax). Наконец, вместо тРНК-подобной D-ножки с укороченной трехнуклеотидной D-петлей, характерной для бактериальных тмРНК, митохондриальные аналоги имеют очень вариабельную петлю длиной от 5 до 14 нуклеотидов. Промежуточная последовательность (Int.) Состоящей из двух частей mt-tmRNAs богата A + U и имеет неправильную длину (4-34 нуклеотида). ). Модели вторичной структуры одно- и двухкомпонентных мт-тмРНК см. На рисунке 1.
Обработка и экспрессия мт-тмРНК [ править ]
Данные РНК-Seq Phytophthora sojae показывают уровень экспрессии, сходный с уровнем экспрессии соседних митохондриальных тРНК , а четыре основных сайта процессинга подтверждают предсказанные концы зрелой мт-тмРНК. [29] Молекула-предшественник тмРНК, вероятно, процессируется РНКазой P и 3'-процессирующей эндонуклеазой тРНК (см. Рис. 2); Предполагается, что последнее действие приведет к удалению промежуточной последовательности. После добавления CCA к 3'-дискриминаторному нуклеотиду тмРНК может заряжаться аланил-тРНК синтетазой с аланином.
См. Также [ править ]
- CLPP
- Рибосома
- Посланник РНК
Ссылки [ править ]
- ^ Keiler KC (2008). «Биология трансляции». Ежегодный обзор микробиологии . 62 : 133–51. DOI : 10.1146 / annurev.micro.62.081307.162948 . PMID 18557701 .
- ^ Ray BK, Apirion D (июль 1979). «Характеристика 10S РНК: новая стабильная молекула РНК из Escherichia coli». Молекулярная и общая генетика . 174 (1): 25–32. DOI : 10.1007 / BF00433301 . PMID 384159 .
- ^ Tyagi JS, Kinger АК (январь 1992). «Идентификация структурного гена РНК 10Sa Mycobacterium tuberculosis» . Исследования нуклеиновых кислот . 20 (1): 138. DOI : 10,1093 / NAR / 20.1.138 . PMC 310338 . PMID 1371186 .
- ^ a b Komine Y, Kitabatake M, Yokogawa T, Nishikawa K, Inokuchi H (сентябрь 1994). «Подобная тРНК структура присутствует в 10Sa РНК, небольшой стабильной РНК из Escherichia coli» . Труды Национальной академии наук Соединенных Штатов Америки . 91 (20): 9223–7. DOI : 10.1073 / pnas.91.20.9223 . PMC 44784 . PMID 7524073 .
- Перейти ↑ Williams KP, Bartel DP (декабрь 1996 г.). «Филогенетический анализ вторичной структуры тмРНК» . РНК . 2 (12): 1306–10. PMC 1369456 . PMID 8972778 .
- ^ Felden В, Himeno Н, Муто А, Маккатчеон ДП, Аткинса ДФ, Gesteland РФ (январь 1997). «Исследование структуры 10Sa РНК (тмРНК) Escherichia coli» . РНК . 3 (1): 89–103. PMC 1369465 . PMID 8990402 .
- ^ a b Zwieb C, Wower I, Wower J (май 1999 г.). «Сравнительный анализ последовательности тмРНК» . Исследования нуклеиновых кислот . 27 (10): 2063–71. DOI : 10.1093 / nar / 27.10.2063 . PMC 148424 . PMID 10219077 .
- ↑ Andersen ES, Lind-Thomsen A, Knudsen B, Kristensen SE, Havgaard JH, Torarinsson E, Larsen N, Zwieb C, Sestoft P, Kjems J, Gorodkin J (ноябрь 2007 г.). «Полуавтоматическое улучшение выравнивания РНК» . РНК . 13 (11): 1850–9. DOI : 10,1261 / rna.215407 . PMC 2040093 . PMID 17804647 .
- ^ a b Gur E, Sauer RT (октябрь 2008 г.). «Эволюция тега деградации ssrA в Mycoplasma: переключение специфичности на другую протеазу» . Труды Национальной академии наук Соединенных Штатов Америки . 105 (42): 16113–8. DOI : 10.1073 / pnas.0808802105 . PMC 2570983 . PMID 18852454 .
- ^ WOWER И.К., Zwieb C, WOWER J (май 2005). «Трансфер-мессенджер РНК разворачивается при прохождении через рибосому» . РНК . 11 (5): 668–73. DOI : 10,1261 / rna.7269305 . PMC 1370753 . PMID 15811920 .
- ^ a b Кейлер К.С., Шапиро Л., Уильямс КП (июль 2000 г.). «тмРНК, которые кодируют индуцирующие протеолиз метки, обнаружены во всех известных бактериальных геномах: двухкомпонентная тмРНК функционирует в Caulobacter» . Труды Национальной академии наук Соединенных Штатов Америки . 97 (14): 7778–83. DOI : 10.1073 / pnas.97.14.7778 . PMC 16621 . PMID 10884408 .
- ^ Sharkady С.М., Williams КП (2004). «Третья линия с двухкомпонентной тмРНК» . Исследования нуклеиновых кислот . 32 (15): 4531–8. DOI : 10.1093 / NAR / gkh795 . PMC 516066 . PMID 15326226 .
- ^ a b c d Джейкоб И., Сейф Э., Паке П.О., Ланг Б.Ф. (апрель 2004 г.). «Потеря мРНК-подобной области в митохондриальных тмРНК якобид» . РНК . 10 (4): 605–14. DOI : 10,1261 / rna.5227904 . PMC 1370551 . PMID 15037770 .
- ^ Шривастава RA, Шривастава N, Apirion D (май 1992). «Характеристика фермента процессинга РНК РНКазы III из клеток дикого типа и сверхэкспрессирующих клеток Escherichia coli при обработке природных субстратов РНК». Международный журнал биохимии . 24 (5): 737–49. DOI : 10.1016 / 0020-711X (92) 90007-N . PMID 1375563 .
- ↑ Li Z, Pandit S, Deutscher MP (март 1998 г.). «Экзорибонуклеолитическое обрезание 3 'является обычным признаком созревания малых стабильных РНК у Escherichia coli» . Труды Национальной академии наук Соединенных Штатов Америки . 95 (6): 2856–61. DOI : 10.1073 / pnas.95.6.2856 . PMC 19659 . PMID 9501180 .
- ^ Мао С, Бхардвай К, Sharkady С.М., Рыба Р.И., Дрискол Т, WOWER Дж, Zwieb С, Собрал BW, Williams КП (2009). «Вариации гена тмРНК» . Биология РНК . 6 (4): 355–61. DOI : 10,4161 / rna.6.4.9172 . PMID 19617710 .
- ^ Someya T, Nameki N, Hosoi H, Suzuki S, Hatanaka H, Fujii M, Terada T, Shirouzu M, Inoue Y, Shibata T, Kuramitsu S, Yokoyama S, Kawai G (январь 2003 г.). «Структура раствора тмРНК-связывающего белка, SmpB, из Thermus thermophilus» . Письма FEBS . 535 (1–3): 94–100. DOI : 10.1016 / S0014-5793 (02) 03880-2 . PMID 12560085 .
- ^ a b Бесшо Ю., Сибата Р., Секин С., Мураяма К., Хигасидзима К., Хори-Такемото С., Широузу М., Курамицу С., Йокояма С. (май 2007 г.). «Структурная основа для функциональной мимикрии тРНК с длинным переменным плечом с помощью РНК-мессенджера» . Труды Национальной академии наук Соединенных Штатов Америки . 104 (20): 8293–8. DOI : 10.1073 / pnas.0700402104 . PMC 1895943 . PMID 17488812 .
- Перейти ↑ Valle M, Gillet R, Kaur S, Henne A, Ramakrishnan V, Frank J (апрель 2003 г.). «Визуализация входа тмРНК в остановившуюся рибосому». Наука . 300 (5616): 127–30. DOI : 10.1126 / science.1081798 . PMID 12677067 .
- ↑ Tu GF, Reid GE, Zhang JG, Moritz RL, Simpson RJ (апрель 1995 г.). «С-концевое удлинение усеченных рекомбинантных белков в Escherichia coli с декапептидом 10Sa РНК» . Журнал биологической химии . 270 (16): 9322–6. DOI : 10.1074 / jbc.270.16.9322 . PMID 7536743 .
- ^ Keiler KC, Уоллер PR, Sauer RT (февраль 1996). «Роль системы маркировки пептидов в деградации белков, синтезированных из поврежденной информационной РНК». Наука . 271 (5251): 990–3. DOI : 10.1126 / science.271.5251.990 . PMID 8584937 .
- ^ Thibonnier M, Thiberge JM, De Повторное использование H (2008). Ахмед Н (ред.). «Транс-трансляция в Helicobacter pylori: необходимость спасения рибосом и необходимость маркировки белков для устойчивости к стрессу и компетентности» . PLOS ONE . 3 (11): e3810. DOI : 10.1371 / journal.pone.0003810 . PMC 2584231 . PMID 19043582 .
- ^ Кирби JE, Trempy JE, Готтесман S (апрель 1994). «Удаление P4-подобного криптического профага приводит к экспрессии протеазы Alp в Escherichia coli» . Журнал бактериологии . 176 (7): 2068–81. DOI : 10.1128 / jb.176.7.2068-2081.1994 . PMC 205313 . PMID 7511583 .
- Перейти ↑ Williams KP (январь 2002 г.). «Веб-сайт тмРНК: вторжение интроном» . Исследования нуклеиновых кислот . 30 (1): 179–82. DOI : 10.1093 / NAR / 30.1.179 . PMC 99078 . PMID 11752287 .
- Перейти ↑ Dwyer DS (январь 2001 г.). «Эгоистичная ДНК и происхождение генов». Наука . 291 (5502): 252–3. DOI : 10.1126 / science.291.5502.252 . PMID 11253208 .
- ^ Уильямс КП (февраль 2003 г.). «Трафик в гене тмРНК» . Журнал бактериологии . 185 (3): 1059–70. DOI : 10.1128 / JB.185.3.1059-1070.2003 . PMC 142792 . PMID 12533482 .
- ^ Hatfull GF, Pedulla ML, Jacobs-Sera D, Cichon PM, Foley A, Ford ME, Gonda RM, Houtz JM, Hryckowian AJ, Kelchner VA, Namburi S, Pajcini KV, Popovich MG, Schleicher DT, Simanek BZ, Smith AL , Зданович Г.М., Кумар В., Пиблз К.Л., Джейкобс В.Р., Лоуренс Дж. Г., Хендрикс Р. В. (июнь 2006 г.). «Изучение метапротеома микобактериофага: геномика фага как образовательная платформа» . PLoS Genetics . 2 (6): e92. DOI : 10.1371 / journal.pgen.0020092 . PMC 1475703 . PMID 16789831 .
- ^ a b Burger G, Gray MW, Забудьте L, Lang BF (2013). «Поразительно бактериоподобные и богатые генами митохондриальные геномы у протистов якобидов» . Геномная биология и эволюция . 5 (2): 418–38. DOI : 10.1093 / GbE / evt008 . PMC 3590771 . PMID 23335123 .
- ^ а б в г Хафез М, Бургер Г, Штейнберг С.В., Ланг Б.Ф. (июль 2013 г.). «Вторая эукариотическая группа с тмРНК, кодируемой митохондриями: идентификация in silico и экспериментальное подтверждение» . Биология РНК . 10 (7): 1117–24. DOI : 10,4161 / rna.25376 . PMC 3849159 . PMID 23823571 . Архивировано из оригинала на 2014-02-21 . Проверено 13 февраля 2014 .
- ^ Эдди, С. "Адский сайт" . Дата обращения 14 августа 2016 .
- Перейти ↑ Eddy SR, Durbin R (июнь 1994). «Анализ последовательности РНК с использованием ковариационных моделей» . Исследования нуклеиновых кислот . 22 (11): 2079–88. DOI : 10.1093 / nar / 22.11.2079 . PMC 308124 . PMID 8029015 .
- ^ Навроцкий EP, Кольбе DL, Eddy SR (май 2009). «Infernal 1.0: вывод выравнивания РНК» . Биоинформатика . 25 (10): 1335–7. DOI : 10.1093 / биоинформатики / btp157 . PMC 2732312 . PMID 19307242 .
Дальнейшее чтение [ править ]
- Хонг С.Дж., Тран QA, Кейлер К.С. (июль 2005 г.). «Регулируемая клеточным циклом деградация тмРНК контролируется РНКазой R и SmpB» . Молекулярная микробиология . 57 (2): 565–75. DOI : 10.1111 / j.1365-2958.2005.04709.x . PMC 3776457 . PMID 15978085 .
Внешние ссылки [ править ]
- tmRDB: база данных последовательностей тмРНК
- Сайт тмРНК
- Запись rfam для тмРНК [ постоянная мертвая ссылка ]